
Новая техника кодирования позволяет «печатать» и хранить данные на ДНК, как старый пишущий станок. Эпигенетическая система маркировки записывает данные в двоичном коде, а нанопоровое устройство секвенирования считывает их результаты. Несмотря на кажущуюся сложность конструкции, этот метод не требует специальных знаний в области лабораторных технологий.
Несмотря на современные достижения в области хранения данных, всё ещё далеко от того, чтобы сравниться с ДНК. Концентрируя огромные объемы информации в крошечных объемах, один кубический сантиметр ДНК может хранить эквивалент 10 эксабайт (ЭБ) данных в течение тысяч лет или даже миллионов при наилучших условиях. Благодаря такому огромному потенциалу ДНК изучается как новый носитель для хранения цифровой информации.
Новейшие технологии позволяют переводить информацию в двоичном формате в последовательности нуклеотидов: аденин, гуанин, цитозин и тимин. Запись происходит путем синтеза молекулы ДНК с чистого листа — добавлением нуклеотидных оснований по заранее заданному алгоритму.
Для преодоления этих ограничений предложили альтернативные методы, например, параллельную запись. Но структурная неустойчивость и низкая производительность обработки информации мешают их использованию. В отличие от этого, эпигеном может кодировать изменчивую информацию, более устойчивую по сравнению с геномной последовательностью, ее несущей. В исследовании под руководством Пекинского университета разработана методика, вдохновленная древними техниками печати и основанная на передаче эпигеномных признаков.
«Вдохновленные естественным эпигетическим наследием и синтетической самосборкой ДНК, разработали нетрадиционную систему записи данных на ДНК. Такая система позволяет параллельно записывать произвольные биты эпигенетической информации (эпи-биты) на шаблоны ДНК с помощью ферментативного метилирования под руководством самосборки ДНК. — поясняют исследователи в статье, изданной в этом журнале. .
Система, созданная по образцу классических печатающих машин.
Методика предусматривает кодирование бинарной информации в ДНК путем селективного метилирования — эпигенетического изменения. Метилированное основание представлено цифрой 1, а неметилированное — цифрой 0. Содержание информации не предоставляет сама по себе последовательность ДНК, а слой метилирования. — поясняет соавтор исследования Лонг Цянь из Пекинского университета в журнале «Chemical & Engineering News».
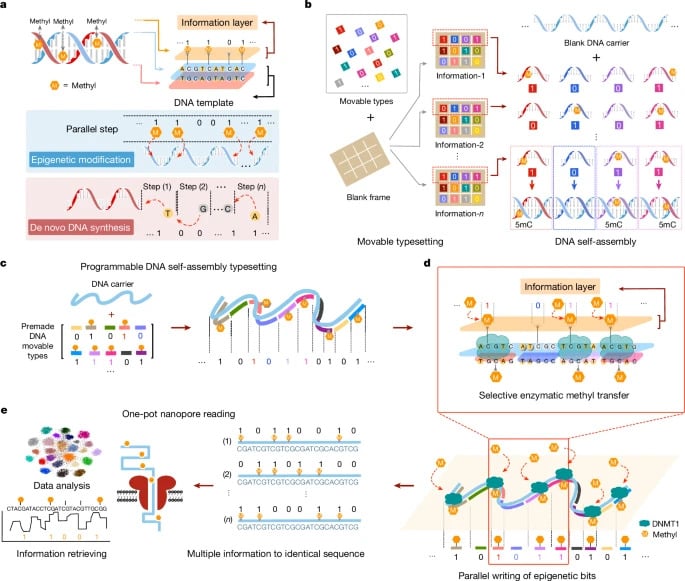
Учёные создали программируемый способ печати с использованием самосборки, напоминающий подвижный шрифт старых типографий. Для печати газет раньше собирали по одному на больших пластинах, формируя текст для печати, а затем покрывали их чернилами, пропуская сквозь них бумагу. В разработанной технике роль бумаги выполняет универсальный шаблон одноцепочечной ДНК, а печатный шрифт представляет собой набор из 700 коротких нитей, называемых «кирпичиками». Каждый кирпичик состоит из 24 нуклеотидов, а цитозины служат местами метилирования.
Информацию кодируют, упорядочивая кирпичики ДНК в заданной последовательности, каждая из которых вмещает эпибитную информацию. Метилтрансфераза (фермент) обрабатывает ДНК, распознает метилированный цитозин и записывает данные о метилировании, взаимодействуя с шаблоном ДНК. Этот процесс аналогичен копированию цифровых данных на жесткие диски путем изменения направления намагничивания. Информацию восстанавливают методом нанопорового секвенирования, позволяющего в реальном времени считывать фрагменты ДНК и состояние их метилирования.
Группа провела испытания своей технологии печати ДНК для записи рисунка тигра и фотографии панды, содержащих 16 833 и 252 500 бит соответственно. Все данные были успешно напечатаны и восстановлены. В отличие от прежних методов, где добавление нуклеотидов занимало от 4 до 20 минут, этот прогресс позволяет одновременно печатать 350 бит информации гораздо быстрее существующих способов.
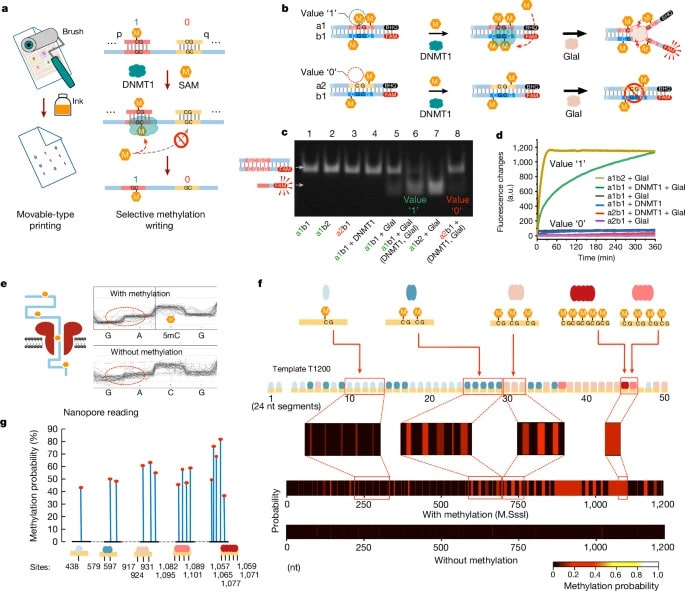
б. Дизайн однобитового анализа записи метилирования.
в. Анализ сдвига геля для выявления различных состояний метилирования.
г. Флуоресцентная детекция различных состояний метилирования.
д. Иллюстрация эпи-битового считывания при нанопоровом секвенировании.
е. Вызов метилирования по сигналам модельной нанопоры.
Доступность для неспециалистов и точность 98,58 %
Исследователи пригласили 60 добровольцев, не имеющих опыта работы с лабораторными методами, чтобы оценить доступность методики. Участникам предложено было воспользоваться платформой для резервного копирования и восстановления данных, а также использовать программное обеспечение под названием iDNAdrive для кодирования текстов общим объемом около 5 000 бит. В то время как хранение нуклеотидов осуществляется на специализированной платформе для синтеза и требует развитых лабораторных навыков, манипуляции для новой техники сводятся к добавлению различных реагентов в одну пробирку. Данные были распечатаны и считаны с точностью 98,58%.
Исследователи планируют продолжать совершенствовать методику, уделяя особое внимание увеличению скорости записи данных и снижению затрат. Также они надеются увеличить объем памяти, распространив эпигенетическую маркировку на другие нуклеотидные основания, а не ограничиваясь только цитозином. В конечном счете, эта технология может произвести революцию в области хранения данных, не в последнюю очередь благодаря своей доступности.